這一篇與[day 26]的差異是,我們對於對於訓練模型上參數的調整。
可以先看一下原始資料集,是之前的kaggle比賽:
https://www.kaggle.com/c/stanford-covid-vaccine/data
主要是想預測對於不同的mRNA片段在不同環境之下裂解的程度(酸鹼度PH10或是50℃ 各自有沒有加鎂離子的情況)。
原始資料還需要考量到mRNA片段結構上的影響,處理起來相對較複雜。
而這裡我們使用QLattice官網教學提供的簡化資料集,
只需要考量不同核苷酸的比例以及各自配對的數量,
來試著預測mRNA片段在酸鹼度PH10加入鎂離子的裂解程度。
https://docs.abzu.ai/docs/tutorials/archive/covid_simple.html

安裝及載入套件:
可以先看一下資料結構,
A、G、C、U為mRNA構成的四種核甘酸,
核甘酸與核甘酸鍵結有U-G、C-G、U-A、G-C、A-U、G-U這幾種情況,
pairs_rate代表了有配對的核甘酸比例,
而mean_deg_Mg_pH10為要預測的標籤 mRNA的裂解程度。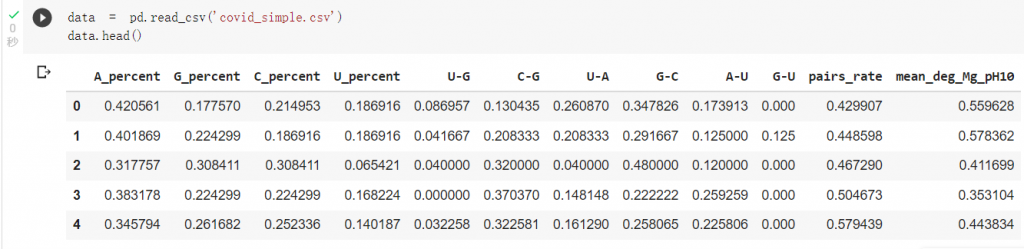
mRNA結構示意圖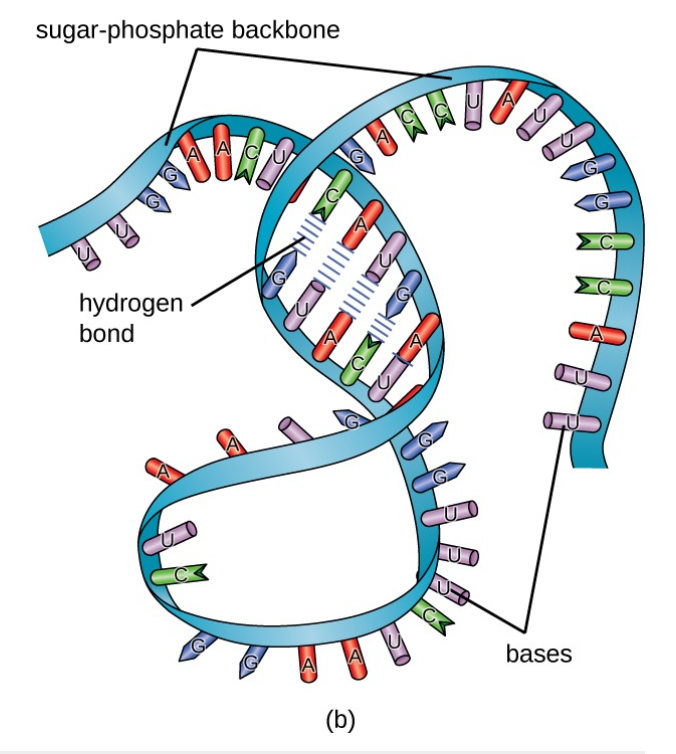
資料來源:
https://courses.lumenlearning.com/microbiology/chapter/structure-and-function-of-rna/
切分訓練集、測試集、驗證集:
我們使用預設的參數開始訓練: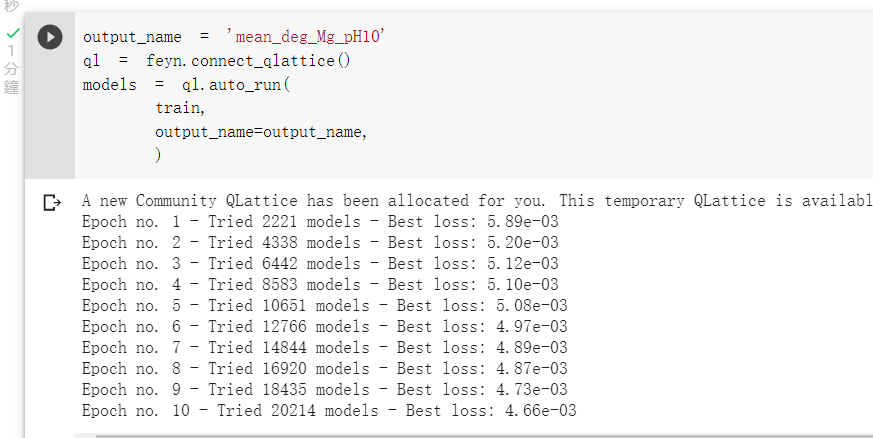
我們可以查看訓練出的最佳模型: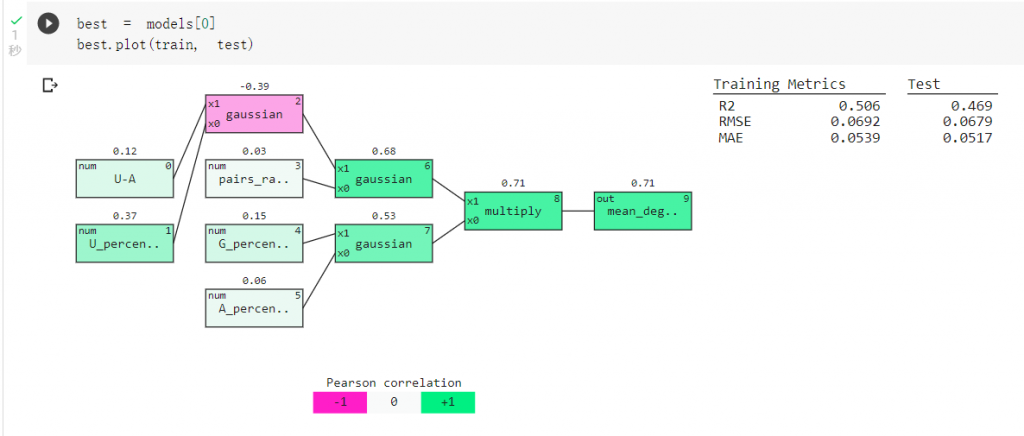
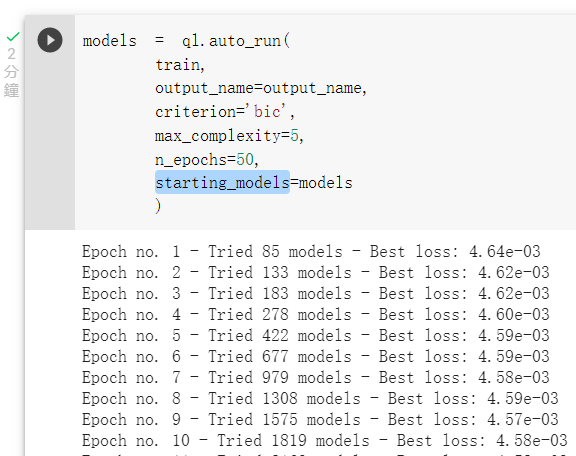
接著我們更改參數
max_complexity: 模型的複雜程度,用4或5可以生成樹狀結構較簡單的模型,
10以上的話會生成較複雜的模型。
n_epochs: 迭代次數,預設是10,越高代表生成模型及淘汰模型次數變高。
criterion: 擬合優化標準有Akaike’s Information Criteria及Bayesian Information Criteria及不選的預 設參數的這幾種選擇(有興趣可以自行調查他們之間的差異),參數名稱分別為'aic'、'bic',或是不給這個參數。
starting_models: 將預設參數訓練完成的模型,接續著訓練。
我們可以查看訓練出的模型,分支變少了: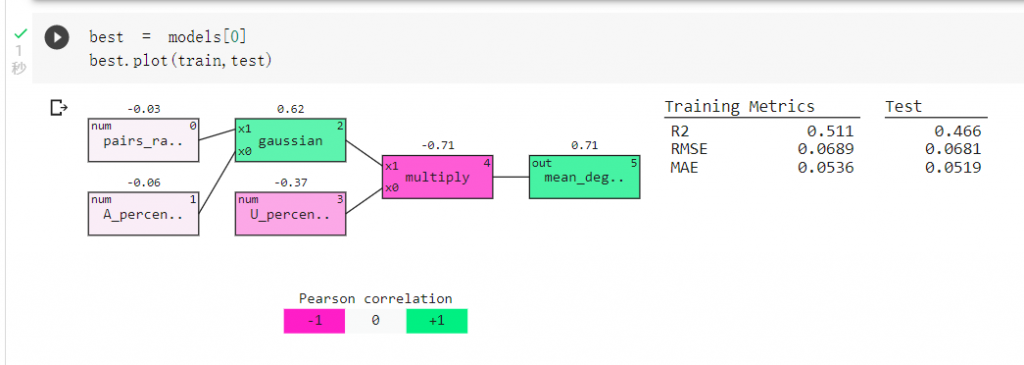
可以發現影響mRNA會不會裂解最主要的因素為核甘酸A、U的比例及核甘酸配對比例。
